前言
2023年,安序源與美國布朗大學Peter Belenky教授課題組(以下簡稱“課題組”)開展基於納米孔基因測序技術的科研合作。
在順利交付安序源AxiLona AXP-100平台後,課題組持續利用該平台進行微生物領域的測試。2024年7月,課題組在微生物組研究期刊Frontiers in Microbiomes上發表了研究論文。
16S rRNA全長1.5kb,傳統二代測序因讀長較短(50-150bp)僅能對部分區段設計引物擴增並測序。課題組基於測試數據,認為高精度的長讀長測序可以輕鬆測通16S rRNA全長序列,提供更高分辨率的菌群分析能力,使得複雜樣本(如腸道樣本)的分析結果更簡單、更可信。
概述
腸道微生物組的研究,尤其是其對於宿主的影響和互相作用、以及在不同環境暴露下的調節,一直是眾多科研工作者關注的焦點。隨着測序技術的成熟和發展,研究者能在種屬水平對微生物種群進行深入的分析,從而探究不同因素影響下微生物的構成及其適應、進化的過程,以及對宿主造成的影響。
文章闡述了利用16S rRNA測序與生物信息學分析來探究飲食、性別及早期環境對阿莫西林暴露下小鼠微生物組的干擾及其恢復情況。研究團隊在傳統短讀長測序方法外,對安序源AxiLona AXP-100長讀長測序方法的結果進行了分析與比較,揭示了高精度長讀長測序對微生物種群分析的應用潛力。

布朗大學Peter Belenky教授課題組與安序源應用支持團隊
方法與結論
研究團隊選取了Jackson Laboratory來源的雄性(MJ)和雌性 (FJ)C57BL/6 小鼠,以及Charles River Laboratories來源的雌性(FC)小鼠,分組進行高糖、高脂肪(西式)飲食和標準高纖維飼料(普通飼料)飲食喂養,通過對糞便樣本進行16S rRNA 測序和生物信息學分析,研究微生物組對抗生素的反應。
通過測序與分析,研究團隊指出,每組小鼠的微生物組構成在使用抗生素前就有不同的起始狀態,這取決於供應商、性別和飲食。而抗生素的使用進一步加劇了這些差異,也表明每組小鼠對這種干擾的反應各不相同。研究團隊發現,與使用抗生素的普通飼料組相比,西式飲食下的微生物組對抗生素的反應更為強烈,其 α 多樣性、β 多樣性以及微生物組成的變化更大。研究團隊認為,這些發現強調了在研究抗生素對小鼠的影響時,考慮供應商來源、飲食和性別因素的重要性。同時指出,在開發人類治療方法時,很可能需要採用基於患者特定微生物組信息的方法,以便在使用抗生素期間保護微生物組。
AxiLona AXP-100測序結果分析
在傳統二代測序技術之外,研究團隊使用安序源的AxiLona AXP-100高精度納米孔測序技術測試了部分樣本並對數據結果進行了分析,以確定抗生素作用於西式飲食組的FJ小鼠後,不同微生物菌種的豐度變化。
研究團隊在第25天這個時間點上,從11個樣本中得到了抗生素暴露組與未暴露組中豐度最高的前 30個科的微生物群落數據。在分析第 25 天這個時間點的3個暴露組樣本和1個未暴露組樣本時,研究團隊觀察到在抗生素暴露條件下,腸桿菌科和阿克曼氏菌科的數量有所增加。在阿克曼氏菌科中,共觀察到兩個菌種,即嗜黏蛋白阿克曼氏菌(Akkermansia muciniphila)和嗜糖阿克曼氏菌(Akkermansia glycaniphila)(圖 5B)。團隊發現,經過抗生素處理後,嗜糖阿克曼氏菌數量減少,而嗜黏蛋白阿克曼氏菌數量增加。在腸桿菌科的63個觀察到的菌種中,一些數量最多的菌種包括大腸桿菌、密歇根克雷伯菌、産酸克雷伯菌和類肺炎克雷伯菌(圖 5C)。
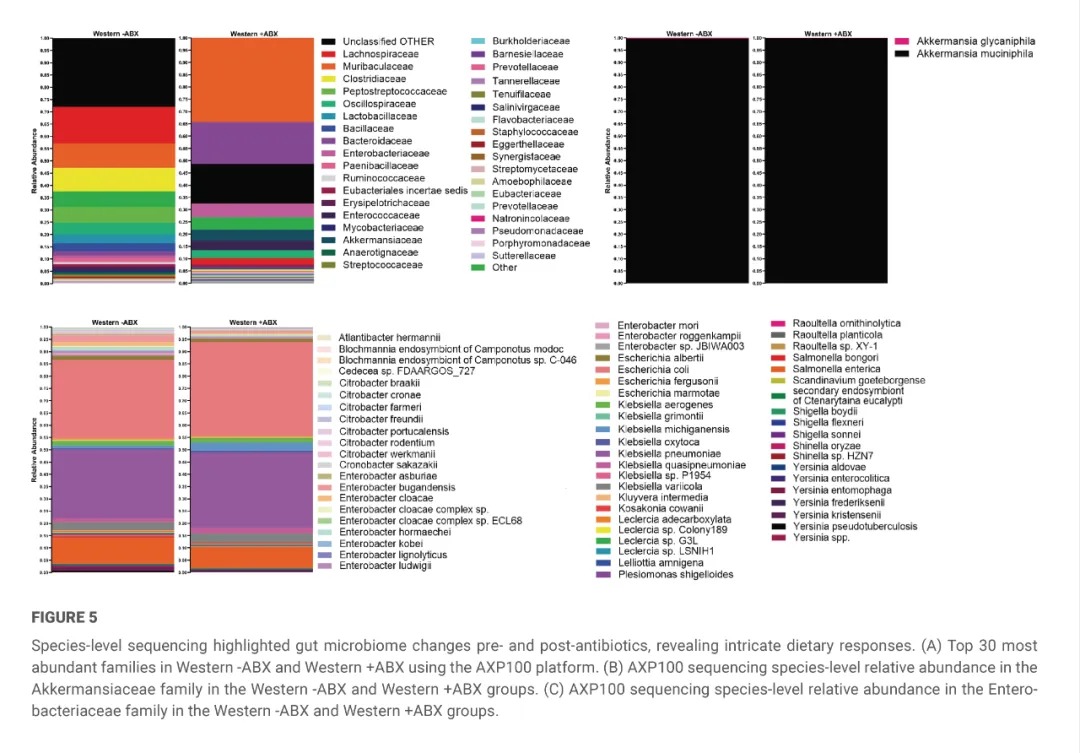
研究團隊認為,儘管這項分析的樣本量(n)不足以開展統計分析,但它確實説明了AxiLona AXP-100長讀長測序技術可用於開展微生物菌種層面的分析。

布朗大學Peter Belenky教授與安序源應用副總裁Nikolay Sergeev博士

